背景
NormAE是由哈尔滨医科大学发表在Analytic Chemistry上的一篇用来去除代谢组学批次效应的深度学习算法。
原文链接

数据准备
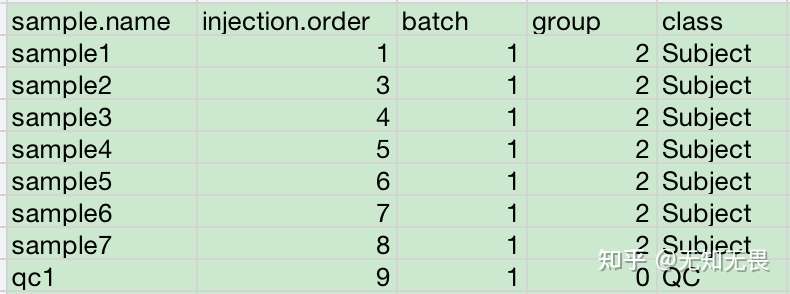
data
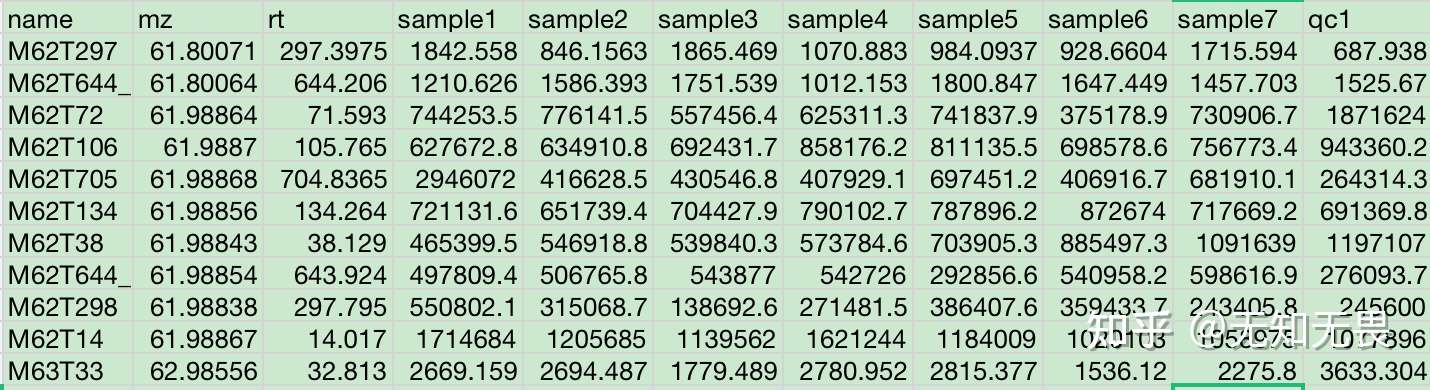
sample.info
环境搭建
- 安装python,以及python上的若干包,列举如下:
python(3.6.8)
pytorch(1.2.0)深度学习框架,用于进行深度学习模型的设计、搭建和训练,其有GPU版本和CPU版本,安装GPU版本将提供额外的GPU加速服务,但需要有Nvidia显卡和相应的cuda c++计算库
numpy(1.17.3)python用于创建和操作矩阵、数组等的标准库
scipy(1.3.1)python用于科学计算的标准库
pandas(0.25.3)可以创建和操作类似R中的dataframe对象,便于进行数据管理
sklearn(0.21.3)机器学习方法
matplotlib(3.1.1)绘图库
visdom(0.1.8.8)一个实时展现数据的包,用于在深度学习训练时动态查看其训练状态
argparse(1.1)设置命令行参数
tqdm(4.32.2)进度条
后面的数字是作者在开发时使用的版本。
克隆代码
在终端中cd到相关文件夹
git clone https://github.com/luyiyun/NormAE.git
训练模型
在命令行环境或终端环境中,进入到NormAE文件夹下,键入以下终端命令
python main.py --task train --meta_data ./DATA/data.csv --sample_data ./DATA/info.csv --save ./RESULTS/train1
当训练结束后,在./RESULTS/train1中将有以下4个文件:
models.pth(储存模型参数的文件)
train.csv(记录了训练时的loss的文件)
config.json(记录了本次训练时使用的超参数和配置)
early_stop_info.json(记录了训练时间和其他的信息)
如果您想使用visdom提供相应的实时可视化,可以重新打开一个命令行或终端窗口,使用
python -m visdom.server
来打开visdom服务,然后在浏览器中输入http://localhost:8097
即可打开visdom界面,如果没有安装visdom,可以在终端输入
pip install visdom -i https://pypi.tuna.tsinghua.edu.cn/simple
移除批次效应
键入以下终端命令
python main.py --task remove --meta_data ./DATA/data.csv --sample_data ./DATA/info.csv --save ./RESULTS/train1
--load ./RESULTS/train1/models.pth
这将使用保存的模型来去除数据中的批次效应。 之后会在./RESULTS/train1增加一下文件:
Ori.csv和Ys.csv(原始的数据集,只是改变的储存方式 Rec.csv(代谢数据的重建,其中还带有批次效应) Codes.csv(NormAE瓶颈层输出的隐含变量值)
Rec_nobe.csv(去除批次效应的重建,我们需要的结果)